即可将网页分享至朋友圈
近日,我校生命科学与技术学院郭锋彪教授课题组在计算分子生物学权威期刊、牛津大学出版社出版的《Nucleic Acids Research》(核酸研究)上发表题为“ZCURVE 3.0: identify prokaryotic genes with higher accuracy as well as automatically and accurately select essential genes”(ZCURVE 3.0:更加准确识别原核生物基因及自动化、准确地选择必需基因)的研究论文。该文目前已在线发表于期刊网站,并将于今年7月1日正式刊登于该刊Web Server专辑。
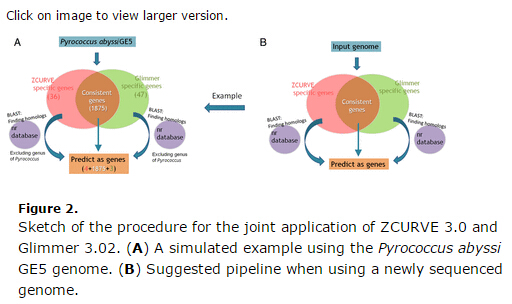
该项工作由我校和天津大学合作完成,由我校信息医学研究中心和大数据研究中心联合资助,受到国家自然科学基金和四川省青年科技基金的支持。即将毕业的生命学院硕士研究生华志刚与天津大学青年教师林岩博士为联合第一作者,指导老师郭锋彪教授为通讯作者,电子科技大学为第一作者单位和通讯作者单位。
在研究中,课题组基于我国生物信息学家、天津大学张春霆院士提出的“Z曲线理论”,改进了细菌基因识别软件ZCURVE,升级后版本为3.0。新版本中,增加了识别变量,将老版本中的Fisher判别替换为更为广泛使用的支持向量机(SVM)分类模型。尤为重要的是,该软件首次提供必需基因的自动化注释,丰富了细菌基因注释工具的功能。研究人员对ZCURVE3.0软件的准确率进行了系统评价,发现新版本的性能不仅远高于老版本,而且和国际上使用最多的细菌基因识别软件Glimmer3.02准确率相当。
该工作属于生物学和信息学交叉的典型工作,是近年来我校在信息学与生物医学交叉研究领域取得的又一科研成果。《Nucleic Acids Research》(《核酸研究》)是牛津大学出版社出版的生化与分子生物学领域权威性期刊,最新影响因子为8.808,5年影响因子为8.378,在JCR期刊分区中,属于生物领域的TOP期刊。
论文链接:
http://nar.oxfordjournals.org/content/early/2015/05/14/nar.gkv491.long
编辑:林坤 / 审核:罗莎 / 发布:一戈


